近日,匹兹堡大学药物遗传研究中心的杨达和张敏课题组借力于一种名为弹性网络回归(Elastic Net regression)的机器学习模型,从1,001个肿瘤细胞系的高通量长非编码RNA表达谱(long noncoding RNAs, lncRNAs)与265种抗癌药物敏感性数据中,挖掘出了27,341对lncRNA-药物可预关联(lncRNA-drug predictive pairs)并绘制出了lncRNA癌症药物基因组图谱。
研究结果以“Systematic Identification of Non-coding pharmacogenomic landscape in cancer”为题发表在《Nature Communications》杂志上。

LncRNA是一类长度超过200个核苷酸的ncRNA,少部分lncRNA在肿瘤细胞生长、迁移及耐药发生中的关键调控作用已被多次报道1-7,但人们对于大部分lncRNA在肿瘤细胞中的调控作用仍缺少系统的认识。近年来,许多研究致力于对上千肿瘤细胞系进行高通量测序与药敏测试,促进了人们对肿瘤耐药机制的认识。然而,这些研究也因局限于蛋白质编码基因而难以完备地阐明耐药的发生与调控8-12。
在该项研究中,研究人员分析了由弹性网络回归模型筛选出的lncRNA-药物可预关联,发现有一部分lncRNA与同种机制多种药物的药敏高度相关,而另一部分lncRNA则与不同机制多种药物的耐药有关。研究人员进一步实验验证了lncRNA EPIC1对BET 蛋白抑制剂在乳腺癌细胞中的耐药调控,并通过转录组测序分析发现它对耐药的调控与MYC通路的激活具有显著相关性。BET抑制剂(Bromodomain and Extra-Terminal motif Inhibitors)是最近发现的一类非常热门的抗癌小分子药物。目前在多个癌症的临床实验中效果都不错。BET抑制剂的抗癌作用的主要机制之一是通过抑制癌基因MYC的表达。
在杨达组于今年4月发表于Cancer Cell上的文章中9,本论文的作者之一王泽华博士证实了EPIC1可直接结合MYC并激活MYC靶基因的转录。结合本文的发现,EPIC1很可能通过提高MYC的转录活性来拮抗BET抑制剂引起的MYC表达抑制从而导致耐药性。此外,研究人员对与不同机制多种药物的药敏都高度相关的lncRNA也进行了进一步分析,他们推测这些lncRNA可能与药物本身在细胞中的代谢有潜在关联。
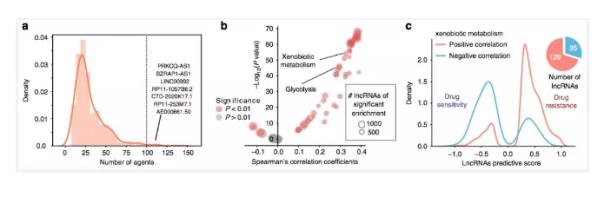
研发药物的成本一直居高临下,因此,人们一直以来都希望通过成本更低、耗时更短的细胞系药敏筛选来建立预测模型,能够在药物进入临床实验前预测癌症病人对药物的药敏性。在本研究中,研究人员对5,605个TCGA病人样本与505个肿瘤细胞系进行相关性分析,发现lncRNA在两者中的表达谱十分相似。因此,他们利用先前筛选出的lncRNA-药物可预关联,为每一个药物建立了一个药敏预测模型。这些基于lncRNA的模型不仅可以很好地预测细胞系中的药敏数据,在这些病人样本中亦具有很好的预测表现。
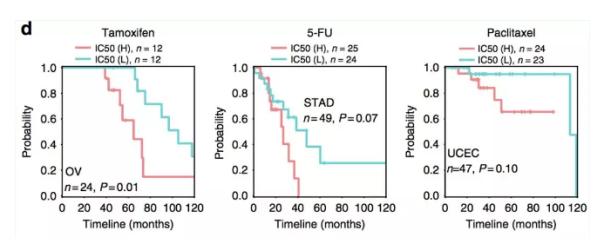
研究人员推测耐药的病人可能具有较差的生存曲线,发现在接受他莫昔芬(tamoxifen)治疗的卵巢癌、5-FU治疗的胃癌及紫杉醇(paclitaxel)治疗的子宫内膜癌患者中,被模型预测为耐药的患者皆具有相对较差的预后,提示了lncRNA在药敏预测中的巨大潜力。
以往测序技术的限制使得许多lncRNA的研究都采取“自下而上”的策略,即先通过大量实验确定lncRNA的功能,再通过lncRNA的功能得出生物学假设它们有可能调控何种药物敏感行。如今由于测序技术的日渐完善,研究人员们拥有了更多大型组学数据的支持,可以从药物出发去寻找与药物存在潜在调控关系的lncRNA,这将为日后基于lncRNA的精准治疗和癌症机制研究提供重要依据。据悉,该论文的第一作者为匹兹堡大学药学院的在读博士生王越。
参考资料:1)Guttman, M. et al. Chromatin signature reveals over a thousand highly conserved large non-coding RNAs in mammals. Nature 458, 223-227, doi:10.1038/nature07672 (2009).
2)Schmitt, A. M. & Chang, H. Y. Long Noncoding RNAs in Cancer Pathways. Cancer Cell 29, 452-463, doi:10.1016/j.ccell.2016.03.010 (2016).
3)Wang, D. et al. LncRNA MALAT1 enhances oncogenic activities of EZH2 in castration-resistant prostate cancer. Oncotarget 6, 41045-41055, doi:10.18632/oncotarget.5728 (2015).
4) Marques Howarth, M. et al. Long noncoding RNA EWSAT1-mediated gene repression facilitates Ewing sarcoma oncogenesis. J Clin Invest 124, 5275-5290, doi:10.1172/JCI72124 (2014).
5) Agrelo, R. et al. SATB1 defines the developmental context for gene silencing by Xist in lymphoma and embryonic cells. Dev Cell 16, 507-516, doi:10.1016/j.devcel.2009.03.006 (2009).
6)Lee, S. et al. Noncoding RNA NORAD Regulates Genomic Stability by Sequestering PUMILIO Proteins. Cell 164, 69-80, doi:10.1016/j.cell.2015.12.017 (2016).
7)Ling, H. et al. CCAT2, a novel noncoding RNA mapping to 8q24, underlies metastatic progression and chromosomal instability in colon cancer. Genome Res 23, 1446-1461, doi:10.1101/gr.152942.112 (2013).
8) Barretina, J. et al. The Cancer Cell Line Encyclopedia enables predictive modelling of anticancer drug sensitivity. Nature 483, 603-607, doi:10.1038/nature11003 (2012).
9) Perou, C. M. et al. Molecular portraits of human breast tumours. Nature 406, 747-752, doi:10.1038/35021093 (2000).
10 )Cancer Genome Atlas, N. Comprehensive molecular portraits of human breast tumours. Nature 490, 61-70, doi:10.1038/nature11412 (2012).
11) Hancock, A. L. et al. A CTCF-binding silencer regulates the imprinted genes AWT1 and WT1-AS and exhibits sequential epigenetic defects during Wilms' tumourigenesis. Hum Mol Genet 16, 343-354, doi:10.1093/hmg/ddl478 (2007).
12)Jiang, P. et al. Genome-Scale Signatures of Gene Interaction from Compound Screens Predict Clinical Efficacy of Targeted Cancer Therapies. Cell Syst 6, 343-354 e345, doi:10.1016/j.cels.2018.01.009 (2018).
13) Wang, Z. et al. lncRNA Epigenetic Landscape Analysis Identifies EPIC1 as an Oncogenic lncRNA that Interacts with MYC and Promotes Cell-Cycle Progression in Cancer. Cancer Cell 33, 706-720 e709, doi:10.1016/j.ccell.2018.03.006 (2018).
